Bioinformatics Department
ΕΡΕΥΝΑ, Βιοπληροφορική κατά SARs-CoV-2

Το Τμήμα Βιοπληροφορικής (ΤΒ) του ΙΝΓΚ ενεπλάκη ενεργά στην προσπάθεια της επιστημονικής κοινότητας στη μελέτη του ιού SARs-CoV-2 και στην αντιμετώπιση της COVID-19 από την αρχή της πανδημίας. Ο SARS-CoV-2 είναι ένα ιός με τεράστιες δυνατότητες μεταλλάξεων (viral quasispecies), επομένως, η ανθρωπότητα αντιμετωπίζει ένα σύνολο με τρισεκατομμύρια διαφορετικές παραλλαγές του ιού. Πολλές μελέτες έχουν διεξαχθεί σε εθνικό επίπεδο από πολλές χώρες, προκειμένου να παραχθούν και να αναλυθούν δεδομένα υψηλής απόδοσης για τα στελέχη SARS-CoV-2. Πρόσφατα, το Ινστιτούτο Νευρολογίας και Γενετικής Κύπρου (ΙΝΓΚ) και συγκεκριμένα το Τμήμα Μοριακής Ιολογίας (ΤΜΙ) με την επικεφαλής καθηγήτρια Χριστίνα Χριστοδούλου και τον Δρ Jan Richter δημοσίευσαν 144 δείγματα NGS SARs-CoV-2 που ελήφθησαν από τον κυπριακό πληθυσμό και αντιπροσωπεύουν έναν από τους πρώτους τεκμηριωμένους γονιδιωματικούς και επιδημιολογικούς χαρακτηρισμούς στελεχών του ιού στην Κύπρο (Richter et al., 2021). Το ΤΒ του ΙΝΓΚ, σε στενή συνεργασία με το ΤΜΙ, υπέβαλε αυτά τα δείγματα από τον κυπριακό πληθυσμό σε εκτενή υπολογιστική ανάλυση χρησιμοποιώντας ένα ευρύ φάσμα στατιστικών αλλά και υπολογιστικών μεθοδολογιών (Oulas, Richter, et al., 2021). Επίσης το ΤΒ εκπόνησε και άλλες μελέτες με στόχο την μοριακή διερεύνηση του ιού καθώς και τον επαναπροσδιορισμό φαρμάκων για την καταπολέμηση του (Oulas, Zanti, et al., 2021; Tomazou et al., 2021).
Αρχικά το ΤΒ πραγματοποίησε βασική γενεαλογική ανάλυση για τον εντοπισμό των κυρίαρχων στελεχών του SARS-CoV-2 στην Κύπρο. Επιπλέον, πραγματοποιήθηκε φυλογενετική ανάλυση για τη σύγκριση των στελεχών που βρέθηκαν στην Κύπρο έναντι άλλων στελεχών που υπάρχουν στον κόσμο (Εικ. 1).
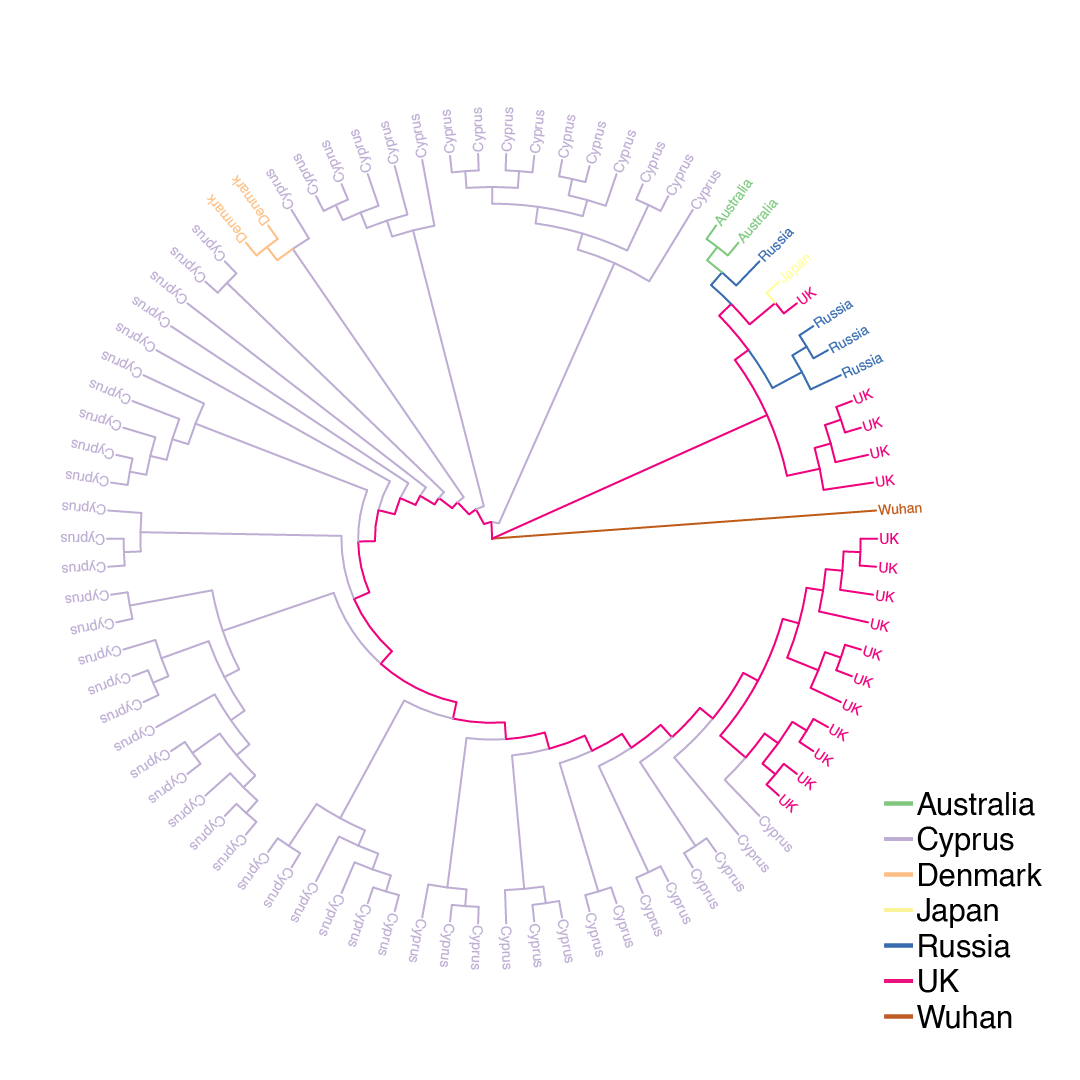
Εικ. 1. Φυλογενετικό δέντρο συγκεκριμένων στελεχών του ιού που βρέθηκαν στην Κύπρο και που φέρουν μεταλλάξεις μοναδικές για τον Κυπριακό πληθυσμό, έναντι άλλων στελεχών που υπάρχουν στον κόσμο. Το στέλεχος αναφοράς Wuhan προστέθηκε για την ολοκλήρωση του φυλογενετικού δέντρου.
Το ΤΒ πραγματοποίησε λεπτομερή γονιδιωματική ανάλυση των κυπριακών δειγμάτων εφαρμόζοντας τεχνικές ανίχνευσης μεταλλάξεων με σκοπό τον προσδιορισμό τής συχνότητας συγκεκριμένων μεταλλάξεων στα στελέχη του ιού που βρέθηκαν στην Κύπρο. Η μελέτη αυτή αποκάλυψε σημαντικές πληροφορίες σχετικά με συγκεκριμένα στελέχη και την συχνότητά τους στον κυπριακό πληθυσμό. Για παράδειγμα, η μελέτη αυτή ανέλυσε τις 9 μεταλλάξεις στην πρωτεΐνη ακίδα (S protein) του ιού που χαρακτηρίζουν την παραλλαγή B.1.1.7 που είχε πρωτοεμφανιστεί στο Ηνωμένο Βασίλειο (Εικ. 2). Στην συνέχεια διερευνήθηκε κατά πόσο η παρουσία του στελέχους αυτού στην Κύπρο, στα μέσα του 2021, επηρέασε τον αριθμό των κρουσμάτων και των θανάτων στον πληθυσμό.
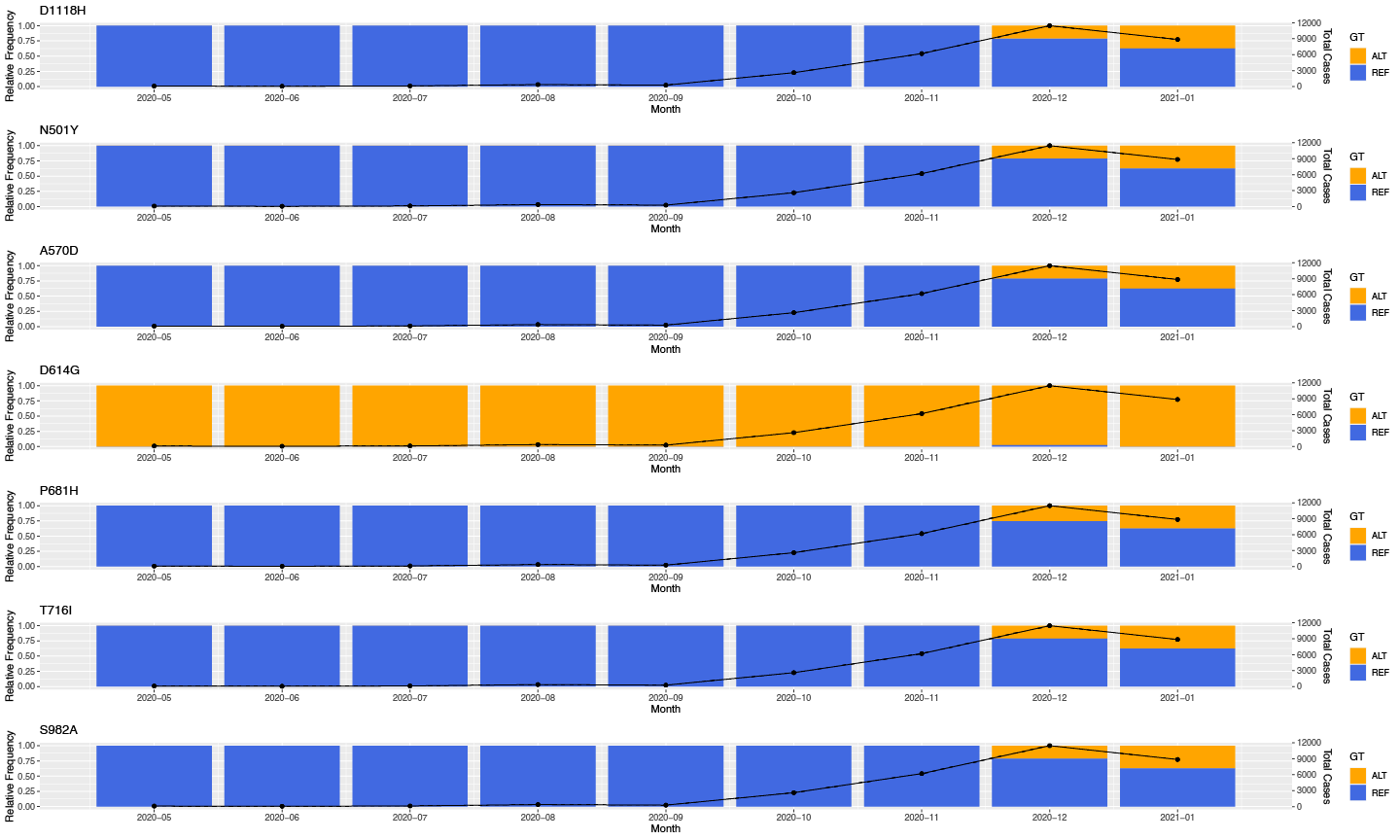
Εικ. 2 Ανίχνευση μεταλλάξεων (7/9) που χαρακτηρίζουν την παραλλαγή B.1.1.7 που είχε πρωτοεμφανιστεί στο Ηνωμένο Βασίλειο. Η εναλλακτική μορφή (ALT) των μεταλλάξεων παρουσιάζεται με πορτοκαλί χρώμα, ενώ η μορφή αναφοράς (REF) των μεταλλάξεων υποδηλώνεται με μπλε χρώμα. Η μαύρη γραμμή δείχνει τον αριθμό των κρουσμάτων ανά μήνα (όπως αναφέρθηκε για την Κύπρο).
Επιπλέον, το ΤΒ εφάρμοσε υπολογιστική ανάλυση μεταλλάξεων προκειμένου να εντοπίσει την προέλευση μεταλλάξεων του ιού που ανιχνεύθηκαν στην Κύπρο, να προσδιορίσει το χρόνο εισαγωγής τους στην χώρα και επίσης να εντοπίσει το βαθμό μετάδοσής τους και διάδοσής τους σε άλλες χώρες. Η μέθοδος αυτή εντόπισε μία μετάλλαξη που βρέθηκε στο γονίδιο ORF1ab η οποία αντιστοιχεί στην πρωτεΐνη NSP14 και φαίνεται να είχε εμπλουτιστεί σημαντικά εντός του κυπριακού πληθυσμού. Το ΤΒ εφάρμοσε υπολογιστικούς αλγορίθμους μηχανικής μάθησης για να προσδιορίσει την επιθετικότητα στελεχών του ιού που φέρουν τη συγκεκριμένη μετάλλαξη.
Μία ακόμα σχετική έρευνα που πραγματοποίησε το ΤΒ σε συνεργασία με τη Μονάδα Βιοστατιστικής του ΙΝΓΚ είχε στόχο τη δομική μοντελοποίηση πρωτεϊνών του ιού τη σύγκριση της δομή της μεταλλαγμένης πρωτεΐνης έναντι της πρωτεΐνης του αρχικού στελέχους. Τα εργαλεία πρόβλεψης πρωτεϊνικής δομής έδειξαν ότι η εξεταζόμενη μετάλλαξη (S6059F) μεταβάλλει σημαντικά τη δομή της ιικής πρωτεΐνης NSP14 επιφέροντας μια προσαρμοστική αλλαγή στο στέλεχος (Εικ. 3).
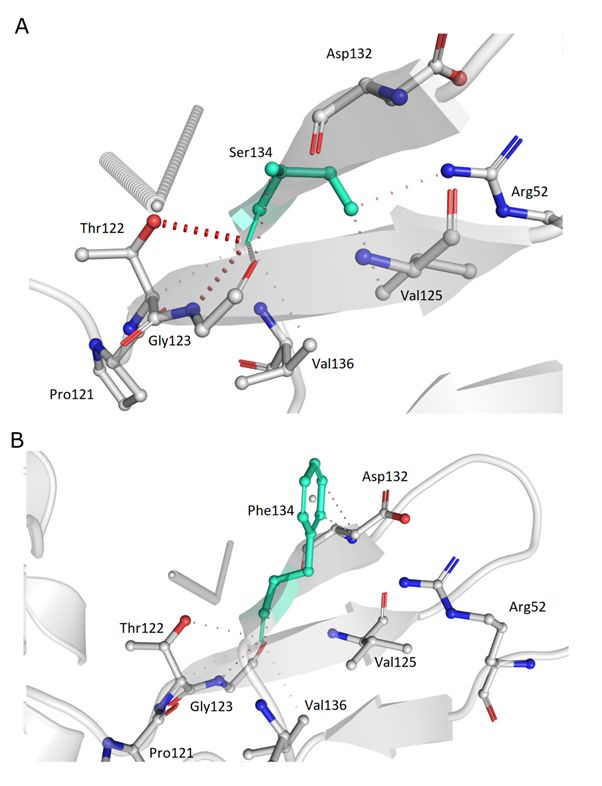
Εικ. 3 Δομικές προβλέψεις της ιικής πρωτεΐνης Α. Μορφή αναφοράς (REF-S6059F). Β. Εναλλακτική μορφή (ALT-S6059F).
Επιπλέον, η μέθοδος αυτή προτείνει ότι η δομική αλλαγή μπορεί να συνδέεται με την υπερμεταλλαξιμότητα του ιού. Βιώσιμες μεταλλάξεις που προκαλούν υπερμεταλλαξιμότητα μπορεί να έχουν πιθανό αντίκτυπο στην δημιουργία νεών στελεχών καθώς και στη συνολική βιωσιμότητα του ιού. Η αυξημένη ικανότητα μεταλλάξεως του γενετικού κώδικα ενός ιού μπορεί να επηρεάσει και την αποτελεσματικότητα των εμβολίων. Μπορεί να υποστηριχθεί ότι οι λοιμώξεις που προκαλούνται από ιούς με τέτοιά υπερμεταλλαξιμότητα μπορούν να λειτουργήσουν ως γεννήτριες διαφορετικών ιογενών στελεχών. Αυτό αυξάνει τις πιθανότητες ανθεκτικότητας του ιού στις τρέχουσες προσπάθειες εμβολιασμού σε σύγκριση με στελέχη με πιο ήπιους ρυθμούς μετάλλαξης.
Συμπερασματικά, η διερεύνηση των μεταλλάξεων για τον SARS-CoV-2 μέσω υπολογιστικών προσεγγίσεων μπορεί να οδηγήσει σε καλύτερη κατανόηση της ιογενούς παθογένειας. Η εφαρμογή μεθόδων βιοπληροφορικής για την ανάλυση αυτών των μεταλλάξεων σε τοπικό επίπεδο μπορούν να αναδείξουν πιθανές συσχετίσεις μεταξύ των ιικών μεταλλάξεων και της μοναδικής γονιδιωματικής του Κυπριακού πληθυσμού. Αυτό μπορεί να οδηγήσει σε σημαντικά ευρήματα για την αντιμετώπιση της πανδημίας τόσο σε εθνικό όσο και σε παγκόσμιο επίπεδο.
Το ΤΒ επίσης καθοδήγησε μια πολύ-επίπεδη έρευνα σε συνεργασία με άλλους επιστήμονες από την Κύπρο (Πανεπιστήμιο Κύπρου και Πανεπιστήμιο Λευκωσίας) και την Ελλάδα (Δημοκρίτειο Πανεπιστήμιο Θράκης) για τον επαναπροσδιορισμό φαρμάκων για την καταπολέμηση της ασθένειας COVID-19 (Tomazou et al., 2021). Η έρευνα αυτή ανέδειξε μια καινοτόμα μέθοδο υπολογιστικής σύνθεσης πολυ-επίπεδων ομικών δεδομένων. Η μελέτη κατέληξε σε σημαντικά γονίδια και μοριακούς μηχανισμούς που σχετίζονται με την COVID-19, αναδεικνύοντας υποψήφια δραστικά φάρμακα για την καταπολέμησή της. Ο στόχος της μελέτης ήταν η ανάπτυξη και εφαρμογή μιας σύνθετης και καινοτόμου μεθοδολογίας ενοποίησης πολύ-επίπεδων δεδομένων βασισμένη σε δίκτυα αναπαράστασης και συσχέτισης δεδομένων (“network-based integration of multi-omic data” – Εικ. 4) για την χαρτογράφηση γνωστών φαρμάκων ως πιθανές θεραπείες έναντι της COVID-19. Οι ερευνητές του ΤΒ μαζί με τους συνεργάτες τους χρησιμοποίησαν και ανέλυσαν διαθέσιμα δεδομένα από πειράματα σε κύτταρα, από ασθενείς και υγιείς ανθρώπους, καθώς και από διεθνείς βάσεις δεδομένων για να συνθέσουν λίστες γνωστών φαρμάκων που σχετίζονται με τη βιολογία, τη γενετική και την παθολογία του ιού SARS-COV-2. Ανάμεσα σε χιλιάδες φάρμακα που αναλύθηκαν, η έρευνα κατέληξε σε μια διαβαθμισμένη λίστα 65 φαρμάκων με τις υψηλότερες προσδοκίες για πιθανή δράση έναντι της ασθένειας COVID-19, από την οποία υπογραμμίστηκαν 16 φάρμακα που παρουσιάζουν ιδιαίτερο ενδιαφέρον για περαιτέρω μελέτες.
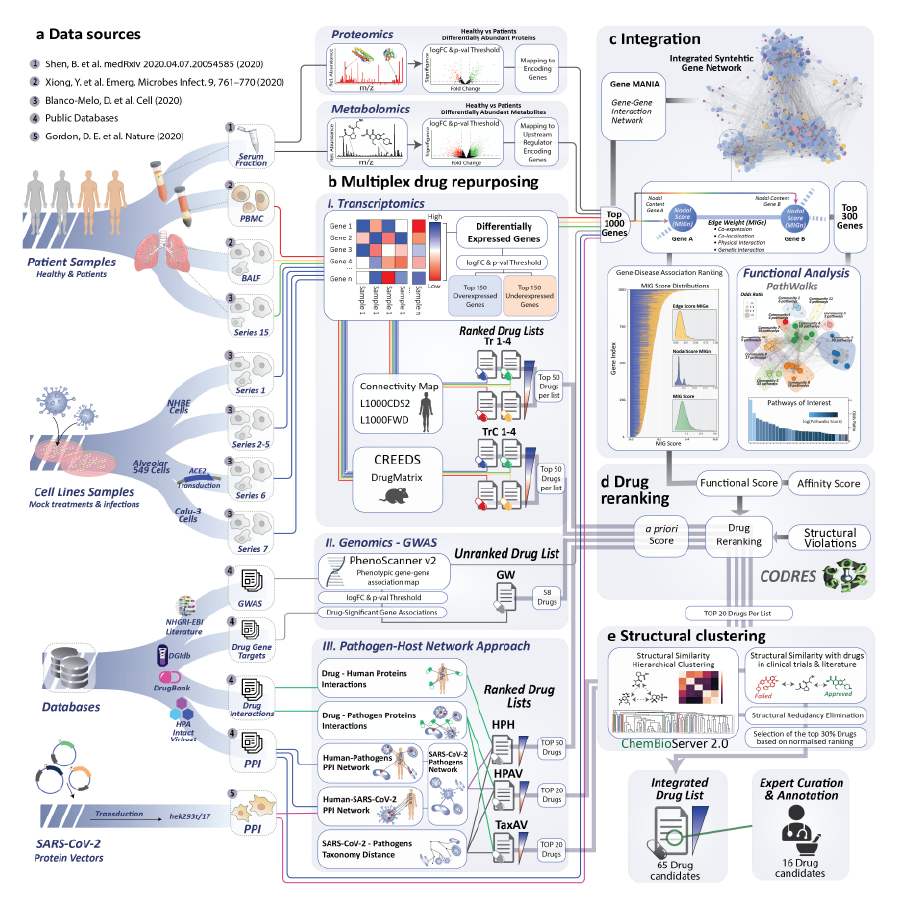
Εικ. 4 Διάγραμμα ροής που περιγράφει τη μεθοδολογία ενοποίησης πολύ-επίπεδων δεδομένων μέσω δικτύων και τις συσχετίσεις για την χαρτογράφηση γνωστών φαρμάκων ως πιθανές θεραπείες έναντι της COVID-19.
Βιβλιογραφία
- Oulas,A., Zanti,M., et al. (2021) Generalized linear models provide a measure of virulence for specific mutations in SARS-cov-2 strains. PLoS One, 16.
- Oulas,A., Richter,J., et al. (2021) In depth analysis of Cyprus-specific mutations of SARS-CoV-2 strains using computational approaches. BMC Genomic Data.
- Richter,J. et al. (2021) Molecular epidemiology of SARS-CoV-2 in Cyprus. PLoS One, 16, e0248792.
- Tomazou,M. et al. (2021) Multi-omics data integration and network-based analysis drives a multiplex drug repurposing approach to a shortlist of candidate drugs against COVID-19. Brief. Bioinform.
Άρθρο
Αναστάσιος Ούλας, PhD
Associate Scientist
Τμήμα Βιοπληροφορικής









0002.jpg)
